> Statistiques > Apprentissage > Arbres de décision (rpart)
Arbres de décision (rpart)
Objectif : prédire une variable en fonction d'attributs pour une liste d'individus. On suppose avoir une liste d'individus caractérisés par des variables explicatives, et on cherche à prédire une variable expliquée. L'apprentissage se fait par partionnement récursif des instances selon des règles sur les variables explicatives. Deux types d'arbres de décision :
- arbres de classification : la variable expliquée est de type nominale (facteur). A chaque étape du partitionnement, on cherche à réduire l'impureté totale des deux noeuds fils par rapport au noeud père.
- arbres de régression : la variable expliquée est de type numérique et il s'agit de prédire une valeur la plus proche possible de la vraie valeur.
Implémentation documentée ici : rpart. Attention, il faut charger la librairie par library(rpart)
Exemple d'utilisation simple avec variable prédite de type facteur (arbre de classification) :
fr <- data.frame(x = runif(1000, 0, 3), y = runif(1000, 2, 5))
fr$z <- factor(ifelse(fr$x < 2, "a", ifelse(fr$y > 4, "b", "a")))
fit <- rpart(z ~ x + y, fr, method = "class")
L'objet retourné est de la classe rpart. L'argument
method="class" est optionnel (automatique par défaut car la variable prédite est de type facteur).
Impression de l'arbre sous forme textuelle pour un arbre de classification :
n= 1000
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 1000 116 a (0.8840000 0.1160000)
2) y< 4.014725 691 0 a (1.0000000 0.0000000) *
3) y>=4.014725 309 116 a (0.6245955 0.3754045)
6) x< 2.003892 193 0 a (1.0000000 0.0000000) *
7) x>=2.003892 116 0 b (0.0000000 1.0000000) *
A chaque noeud, on a :
- Le numéro de noeud : 2) (noeuds de gauche et droite numérotés 2x et 2x + 1 si père numéroté x).
- le critère de split (ou root pour la racine) : y< 4.014725
- le nombre total d'instances pour le noeud : 691
- le nombre d'instances mal classées (0 => toutes les instances sont bien prédites) : 0
- la valeur prédite (donc majoritaire) de la variable à prédire : a
- entre parenthèses, les proportions d'instances bien et mal prédites : (1.0000000 0.0000000)
- une '*' si c'est un noeud terminal.
On peut avoir tout le détail en faisant :
summary(fit).
Exemple d'utilisation avec variable prédite de type numérique (arbre de régression) :
fr <- data.frame(x = runif(1000, 0, 3), y = runif(1000, 2, 5))
fr$z <- ifelse(fr$x < 2, 2 * fr$x, 3 * fr$y)
fit <- rpart(z ~ x + y, fr, method = "anova")
Impression de l'arbre sous forme textuelle pour un arbre de régression :
n= 1000
node), split, n, deviance, yval
* denotes terminal node
1) root 1000 18975.56000 4.807525
2) x< 2.003762 668 889.05200 1.996973
4) x< 1.0084 341 115.48120 1.021080 *
5) x>=1.0084 327 110.15180 3.014648 *
3) x>=2.003762 332 2192.93300 10.462490
6) y< 3.548979 179 326.58590 8.429348
12) y< 2.812628 90 40.22737 7.272919 *
13) y>=2.812628 89 44.28705 9.598771 *
7) y>=3.548979 153 260.75250 12.841140
14) y< 4.306183 81 33.33708 11.754910 *
15) y>=4.306183 72 24.32592 14.063150 *
A chaque noeud, on a :
- le numéro de noeud 2)
- le critère de split (ou root pour la racine) : x < 2.003762
- le nombre total d'instances pour le noeud : 668
- la déviance, c'est à dire la somme des carrés des écarts à la valeur prédite pour les valeurs de toutes les instances du noeud : 889.05200
- la valeur prédite de la variable à prédire : 1.996973
- une '*' si c'est un noeud terminal.
Impression d'un arbre sous forme graphique :
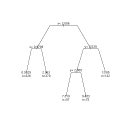
plot(fit, uniform = TRUE, branch = 0.5, margin = 0.1)
text(fit, all = FALSE, use.n = TRUE)
avec les paramètres suivants pour plot (qui dessine l'arbre) :
- uniform : si TRUE, arbre avec arêtes verticales toutes de même longueur, sinon, en fonction de l'erreur liée au split (défaut = FALSE).
- branch : contrôle la forme des branches : 1 si arêtes rectangulaires, 0 si arêtes obliques, entre 0 et 1 pour forme intermédiaire (défaut = 1).
- margin : marge à laisser à l'exterieur de l'arbre pour l'étiquetage (défaut = 0).
et les paramètres suivants pour text (qui étiquette l'arbre) :
- all : FALSE si étiquetage avec la valeur du facteur uniquement pour les noeuds terminaux, TRUE si pour tous les noeuds (défaut = FALSE). Les critères de split sont de toutes façons toujours indiqués.
- use.n : TRUE si indication des effectifs à chaque noeud, FALSE sinon (défaut = FALSE).
Réglage de la complexité de l'arbre : plus l'arbre est complexe (beaucoup de noeuds), plus il va bien apprendre l'échantillon d'apprentissage, mais aussi plus il va faire de l'overfitting : adaptation uniquement à l'échantillon d'apprentissage, mais beaucoup d'erreurs sur un nouvel échantillon de test. La complexité doit donc être pénalisée, d'où un paramètre cp (complexity parameter) :
- plus cp est petit, plus l'arbre peut être grand (beaucoup de noeuds), plus il est grand, plus la complexité est pénalisée.
- contrôle du paramètre de complexité : fit <- rpart(z ~ x + y, fr, method = "class", control = rpart.control(cp = 0.5))
- la valeur par défaut de cp est 0.01
Choix de la complexité de l'arbre :
printcp(fit) affiche pour chaque valeur seuil du paramètre cp :
fr <- data.frame(x = runif(1000, 0, 3), y = runif(1000, 2, 5))
fr$z <- factor(ifelse(jitter(fr$x, amount = 0.5) < 2, "a", ifelse(jitter(fr$y, amount = 0.5) > 4, "b", "a")))
fit <- rpart(z ~ x + y, fr, method = "class", control = rpart.control(cp = 0.00001))
printcp(fit)
affiche :
Root node error: 111/1000 = 0.111
n= 1000
CP nsplit rel error xerror xstd
1 0.342342 0 1.00000 1.00000 0.089493
2 0.018018 2 0.31532 0.44144 0.061499
3 0.006006 4 0.27928 0.36036 0.055827
4 0.000010 7 0.26126 0.39640 0.058430
Pour chaque valeur de cp seuil, on a le nombre de splits de l'arbre correspondant (nsplit), l'erreur croisée xerror et son écart-type xstd. On prend en général la première valeur de cp (i.e. la plus grande) qui est à moins de un écart-type du minimum de xerror. Une fois que la valeur de cp est choisie, on peut récupérer l'arbre correspondant par :
prune(fit, cp = 0.07)
(renvoie un objet de la classe rpart, mais avec certains noeuds supprimés par rapport à l'objet de départ).
Prédiction de valeurs, en sortant une matrice avec les facteurs en colonne et les probabilités des classes en valeurs :
predict(fit, data.frame(x = c(1, 3, 1, 3), y = c(2, 2, 5, 5)))
a b
1 1 0
2 1 0
3 1 0
4 0 1
Prédiction de valeurs, en sortant un vecteur de facteurs :
predict(fit, data.frame(x = c(1, 3, 1, 3), y = c(2, 2, 5, 5)), type = "class")
1 2 3 4
a a a b
Copyright Aymeric Duclert
programmer en R, tutoriel R, graphes en R


